TODO für Tag 11
- GridSearchCV einsetzen
- Beispiel mit SVM-Parametern
GridSearchCV einsetzen
GridSearchCV habe ich ja schon am Tag 9 und Tag 10 verwendet, hier aber noch mal GridSearchCV im Detail. Bei GridSearchCV handelt es sich um ein einfaches Werkzeug, das hilft, die besten Hyperparameter für ein Modell systematisch zu finden. Statt sich auf Intuition oder Trial-and-Error zu verlassen, wird eine Gitter-Suche über alle Kombinationen der angegebenen Parameter durchgeführt. Dabei wird jedes Modell mit Cross-Validation evaluiert, um Überanpassung zu vermeiden und die Verlässlichkeit der Parameterwahl zu erhöhen.
Beispiel:
import pandas as pd
import seaborn as sns
import matplotlib.pyplot as plt
from sklearn.model_selection import GridSearchCV
from sklearn.ensemble import RandomForestClassifier
from sklearn.datasets import load_iris
from sklearn.model_selection import train_test_split
# Beispiel-Datensatz
X, y = load_iris(return_X_y=True)
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.3, random_state=42)
# Parametergrid
param_grid = {
'n_estimators': [50, 100, 200],
'max_depth': [None, 10, 20],
'min_samples_split': [2, 5]
}
# GridSearchCV Setup
grid_search = GridSearchCV(
RandomForestClassifier(random_state=42),
param_grid,
cv=5,
scoring='accuracy',
return_train_score=True
)
grid_search.fit(X_train, y_train)
# Ergebnisse als DataFrame
results = pd.DataFrame(grid_search.cv_results_)
# Beispiel-Plot: Einfluss von max_depth und n_estimators auf den Accuracy-Score
pivot_table = results.pivot_table(
index='param_n_estimators',
columns='param_max_depth',
values='mean_test_score'
)
sns.heatmap(pivot_table, annot=True, fmt=".3f", cmap="YlGnBu")
plt.title("GridSearchCV: Accuracy nach n_estimators und max_depth")
plt.xlabel("max_depth")
plt.ylabel("n_estimators")
plt.tight_layout()
plt.show()GridSearchCV testet alle Kombinationen im param_grid, was bei großen Netzen oder vielen Parametern schnell teuer wird. Alternativ kann man RandomizedSearchCV einsetzen, wenn man die Parameter zufällig testen möchte, aber trotzdem eine gute Lösung erwartet.
Ausgabe:
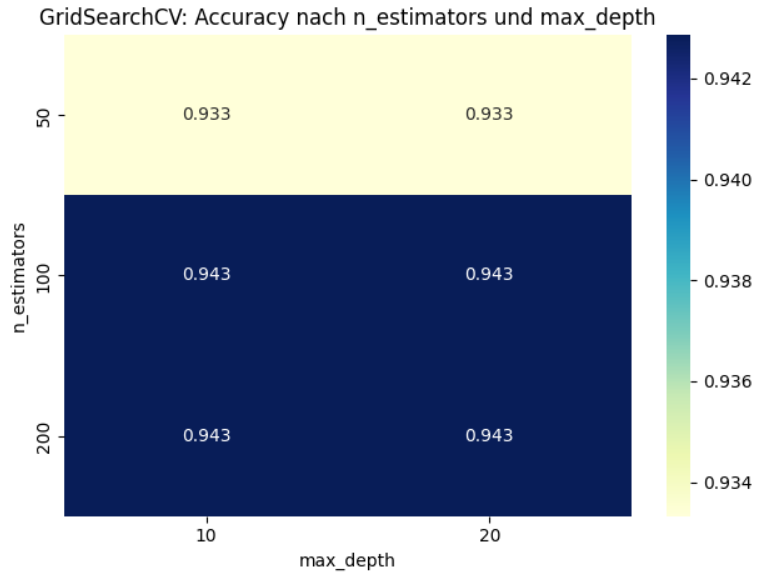
Das Bild visualisiert die die mittlere Cross-Validation-Genauigkeit (mean_test_score) aus dem GridSearchCV-Lauf. Auf den Achsen sind zwei Hyperparameter dargestellt:
- x-Achse:
max_depth(maximale Tiefe der Entscheidungsbäume), - y-Achse:
n_estimators(Anzahl der Bäume im Random Forest).
Bei 50 Bäumen (n_estimators = 50) erreicht das Modell eine mittlere Genauigkeit von 93.3%, unabhängig von der maximalen Tiefe (max_depth = 10 oder 20). Ab 100 Bäumen steigt die Genauigkeit auf 94.3% und bleibt dort auch bei 200 Bäumen stabil – unabhängig von der maximalen Tiefe. Das Modell profitiert sichtbar von einer höheren Anzahl an Entscheidungsbäumen, insbesondere von 50 auf 100. Die Veränderung der max_depth zwischen 10 und 20 hat keinen Einfluss auf die Performance – das spricht dafür, dass entweder die Tiefe kein limitierender Faktor ist, oder die Daten einfach klassifizierbar sind. Mehr als 100 Bäume bringen hier keinen Mehrwert. Man kann sich die längere Trainingszeit bei 200 Bäumen sparen.
Beispiel mit SVM-Parametern
SVC(Support Vector Classification) ist die Implementierung der Support Vector Machine für Klassifikationsaufgaben in scikit-learn. Das Ziel eines SVC-Modells ist es, eine optimale Entscheidungsgrenze zu finden, die die Klassen im Merkmalsraum mit maximalem Abstand trennt. In Kombination mit GridSearchCV kann man gezielt die wichtigsten Hyperparameter des SVC-Modells wie C (Regularisierungsstärke), gamma (Einflussbereich einzelner Punkte im nichtlinearen Raum) und kernel (Art der Trennfunktion) systematisch durchsuchen, um die bestmögliche Modellkonfiguration zu finden. GridSearchCV trainiert dafür alle Kombinationen aus dem angegebenen Parametergrid und bewertet sie mithilfe von Cross-Validation, sodass man objektiv die Einstellungen ermitteln kann, die die beste Generalisierungsleistung liefern.
Beispiel:
from sklearn.model_selection import GridSearchCV
from sklearn.svm import SVC
from sklearn.datasets import load_iris
from sklearn.model_selection import train_test_split
import pandas as pd
# Daten vorbereiten
X, y = load_iris(return_X_y=True)
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.3, random_state=42)
# Parametergrid definieren
param_grid = {
'C': [0.1, 1, 10],
'gamma': ['scale', 0.01, 0.1, 1],
'kernel': ['rbf', 'linear']
}
# SVC mit GridSearchCV
grid_search = GridSearchCV(
SVC(),
param_grid,
cv=5,
scoring='accuracy',
return_train_score=True
)
# Training
grid_search.fit(X_train, y_train)
# Ergebnisse
print("Beste Parameterkombination:")
print(grid_search.best_params_)
print("\nBester Cross-Validation-Score:")
print(f"{grid_search.best_score_:.4f}")
# Optional: Ergebnisse als DataFrame
results_df = pd.DataFrame(grid_search.cv_results_)Dieses Beispiel führt eine systematische Suche nach den besten Hyperparametern für ein SVC-Modell durch, das auf dem Iris-Datensatz trainiert werden soll. Dazu werden zunächst der Iris-Datensatz geladen und in Trainings- und Testdaten aufgeteilt. Anschließend wird ein Gitter möglicher Werte für die SVC-Parameter C, Gamma und Kernel-Typ definiert. Mithilfe von GridSearchCV werden dann alle Kombinationen dieser Parameterwerte auf den Trainingsdaten getestet, wobei die Leistung jeder Kombination durch 5-fache Kreuzvalidierung (cv=5) gemessen wird, um die beste Kombination bezüglich der Klassifikationsgenauigkeit zu finden. Nach Abschluss der Suche werden die Parameterkombination, die den höchsten Kreuzvalidierungs-Score erzielt hat, sowie dieser beste Score selbst ausgegeben.
Ergebnis:
Beste Parameterkombination:
{'C': 1, 'gamma': 'scale', 'kernel': 'linear'}
Bester Cross-Validation-Score:
0.9619
Das Ergebnis zeigt, dass die Suche nach den besten Hyperparametern ergeben hat, dass die Kombination der Parameter C=1, Gamma=’scale‘ und einem linearen Kernel (‚linear‘) die höchste durchschnittliche Genauigkeit erzielte. Der angegebene beste Cross-Validation-Score bedeutet, dass mit dieser Parameterkonfiguration im Durchschnitt über die fünf Validierungsrunden eine Genauigkeit von 96.19% erreicht hat. Dieser sehr hohe Wert deutet darauf hin, dass das gefundene Modell sehr gut darin ist, die Klassen im Iris-Datensatz zu unterscheiden.
Zum Visualisieren kann man auch folgenden Codeschnipsel verwenden.
import seaborn as sns
plt.figure(figsize=(12, 6))
# Auf der x-Achse ist 'C', auf der y-Achse ist die mittlere Test-Genauigkeit
# Verschiedene Linien ('hue='gamma') repräsentieren die verschiedenen Gamma-Werte
g = sns.lineplot(
data=results_df,
x='param_C',
y='mean_test_score',
hue='param_gamma',
marker='o', # Kreise an den Datenpunkten
palette='viridis', # Farbschema
legend='full'
)
plt.tight_layout() # Sorgt für genügend Platz zwischen den Subplots
plt.show()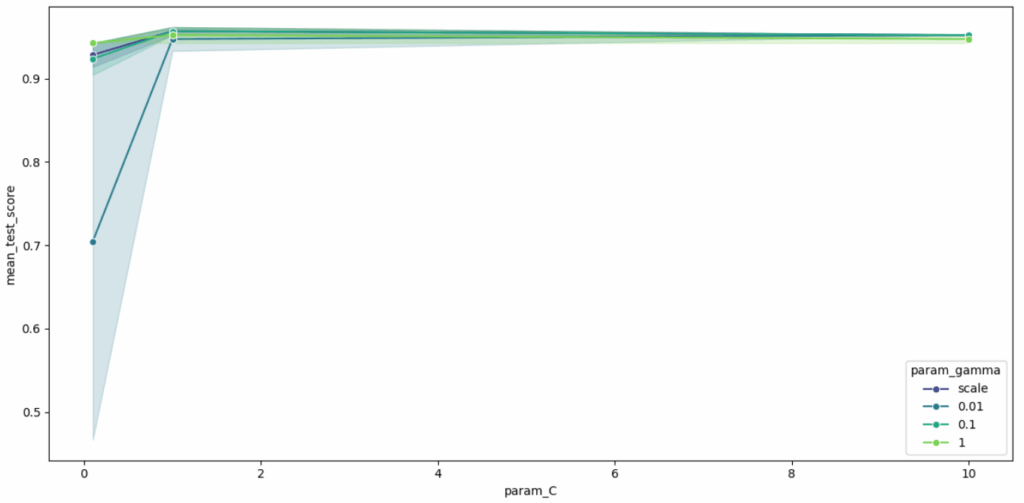
Dieser Plot visualisiert die Ergebnisse der Kreuzvalidierung für das SVC-Modell, indem er die mittlere Test-Genauigkeit (mean_test_score) auf der Y-Achse gegen den Parameter C (param_C) auf der X-Achse aufträgt. Die einzelnen Linien und Farben stellen die verschiedenen getesteten Werte für den Parameter gamma (param_gamma) dar (’scale‘, 0.01, 0.1, 1). Die schattierten Bereiche um die Linien herum zeigen die Variabilität (Standardabweichung) der Genauigkeitswerte über die 5 Folds der Kreuzvalidierung für die jeweilige Parameterkombination.
Der Plot zeigt, dass bei einem kleinen Wert von C (0.1) die Genauigkeit je nach gamma stark variiert und generell niedriger ist. Mit zunehmendem C auf 1 verbessern sich die Genauigkeit für alle gamma-Werte drastisch und erreichen Werte über 0.95, während gleichzeitig die Variabilität über die Folds deutlich abnimmt. Eine weitere Erhöhung von C auf 10 führt zu keiner signifikanten Verbesserung mehr und die Genauigkeit bleibt hoch. Für C Werte von 1 und 10 liefern die getesteten gamma-Werte ’scale‘, 0.1 und 1 sehr ähnliche, hohe Genauigkeiten, was darauf hindeutet, dass das Modell in diesem Bereich robust gegenüber der Wahl von gamma ist, solange C ausreichend groß ist.
Wählt man für C kleinere Werte (0.1, 0.2, 0.3, 0.4,0.5, 0.6, 0.7, 0.8, 0.9, 1) sieht das Ergebnis wie folgt aus:
param_grid = {
'C': [0.1, 0.2, 0.3, 0.4,0.5, 0.6, 0.7, 0.8, 0.9, 1],
'gamma': ['scale', 0.01, 0.1, 1],
'kernel': ['rbf', 'linear']
}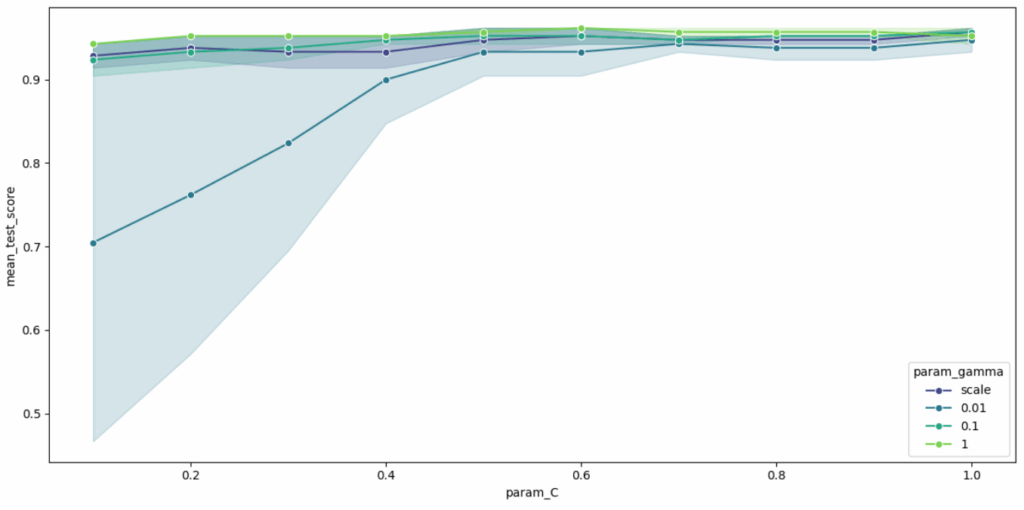
Wie man sieht, zeigt der Plot, wie sich die mittlere Klassifikationsgenauigkeit bei der Kreuzvalidierung ändert, wenn der Parameter C von 0.1 auf 1 erhöht wird. Man sieht, dass bei einem sehr kleinen C (0.1) die Genauigkeit stark vom gamma-Wert abhängt; insbesondere sehr kleine gamma-Werte (wie 0.01 und ’scale‘) führen zu deutlich geringeren und variableren Genauigkeiten als größere gamma-Werte (0.1 und 1). Wenn C erhöht wird, steigen die Genauigkeiten für alle gamma-Werte an und konvergieren ab etwa C=0.5 zu sehr hohen Werten, wobei die Unterschiede zwischen den gamma-Werten geringer werden und die Leistung konsistenter wird (schmalere schattierte Bereiche). Das Bild verdeutlicht somit, dass auf diesem Datensatz ein ausreichender Wert für C (hier ab etwa 0.5) wichtig ist, um eine hohe und stabile Leistung zu erzielen, während bei höheren C-Werten die Wahl des gamma-Werts in diesem Bereich weniger kritisch wird.
Man kann auch eine Pipeline erstellen, bei der der Classifier-Schritt selbst ein Parameter ist, der im GridSearchCV variiert wird. Der param_grid muss dann so strukturiert sein, dass er für jeden Classifier die spezifischen Hyperparameter enthält. In dem folgenden Beispiel werden die Classifier „RandomForestClassifier“, „SCV“ und „LogisticRegression“ mit unterschiedlichen Hyperparametern in einem GridSearch miteinander vergleichen und das Ergebnis ausgegeben.
from sklearn.model_selection import GridSearchCV, train_test_split
from sklearn.ensemble import RandomForestClassifier
from sklearn.svm import SVC
from sklearn.linear_model import LogisticRegression
from sklearn.datasets import make_classification
from sklearn.pipeline import Pipeline
import pandas as pd
import matplotlib.pyplot as plt
import numpy as np # Für np.arange
# Beispieldaten generieren
X, y = make_classification(n_samples=1000, n_features=20, random_state=42)
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.2, random_state=42)
# Pipeline erstellen
pipe = Pipeline([('classifier', SVC())]) # SVC() ist nur ein Platzhalter
# Parameter-Grid definieren
param_grid_pipeline = [
{
'classifier': [RandomForestClassifier(random_state=42)],
'classifier__n_estimators': [50, 100], # Weniger Parameter für schnellere Demo
'classifier__max_depth': [None, 10]
},
{
'classifier': [SVC(probability=True, random_state=42)],
'classifier__C': [0.1, 1, 10],
'classifier__kernel': ['rbf'] # Nur rbf für schnellere Demo
},
{
'classifier': [LogisticRegression(random_state=42, max_iter=200, solver='liblinear')], # solver für Konsistenz
'classifier__C': [0.1, 1, 10],
}
]
# GridSearchCV mit der Pipeline
search = GridSearchCV(pipe, param_grid_pipeline, cv=3, scoring='accuracy', n_jobs=-1, return_train_score=True) # cv=3 für schnellere Demo
search.fit(X_train, y_train)
print(f"Bester Classifier und Parameter: {search.best_params_}")
print(f"Bester Cross-Validation Score: {search.best_score_:.4f}")
# Ergebnisse in einen Pandas DataFrame umwandeln
results_df = pd.DataFrame(search.cv_results_)
# 1. Extrahiere den Namen des Classifiers für jede Zeile
# Die Spalte 'param_classifier' enthält das Classifier-Objekt selbst.
# Wir wollen den Klassennamen als String.
results_df['classifier_name'] = results_df['param_classifier'].apply(lambda x: x.__class__.__name__)
# 2. Finde den besten Score für jeden Classifier-Typ
# Wir gruppieren nach dem Classifier-Namen und nehmen den maximalen 'mean_test_score'
best_scores_per_classifier = results_df.loc[results_df.groupby('classifier_name')['mean_test_score'].idxmax()]
# Daten für den Plot vorbereiten
classifier_names = best_scores_per_classifier['classifier_name']
mean_cv_scores = best_scores_per_classifier['mean_test_score']
std_cv_scores = best_scores_per_classifier['std_test_score'] # Standardabweichung für Fehlerbalken
# --- Plotten der besten Scores pro Classifier ---
plt.figure(figsize=(10, 6))
bars = plt.bar(classifier_names, mean_cv_scores, yerr=std_cv_scores, capsize=5, color=['skyblue', 'lightcoral', 'lightgreen'])
plt.xlabel("Classifier")
plt.ylabel(f"Bester mittlerer CV Score ({search.scoring})")
plt.title("Vergleich der besten Performance verschiedener Classifier")
plt.ylim([min(mean_cv_scores) - 0.05, max(mean_cv_scores) + 0.05]) # Y-Achsen-Limits anpassen
# Werte über den Balken anzeigen
for bar in bars:
yval = bar.get_height()
plt.text(bar.get_x() + bar.get_width()/2.0, yval + 0.005, f'{yval:.3f}', ha='center', va='bottom')
plt.xticks(rotation=15, ha="right")
plt.tight_layout()
plt.show()
# --- Optional: Detailliertere Visualisierung (z.B. Boxplot der Scores für alle Folds pro Classifier) ---
# Dies ist etwas komplexer, da wir die Scores aus den 'splitX_test_score' Spalten extrahieren müssen.
all_scores_data = []
classifier_order_for_boxplot = []
for clf_name in results_df['classifier_name'].unique():
# Finde alle Runs für diesen Classifier
clf_runs = results_df[results_df['classifier_name'] == clf_name]
# Extrahiere die Scores für jeden Fold
# Die Spalten heißen z.B. 'split0_test_score', 'split1_test_score', ...
fold_score_cols = [f'split{i}_test_score' for i in range(search.cv)]
# Sammle alle Fold-Scores für alle Hyperparameter-Kombinationen dieses Classifiers
# Wir nehmen hier nur die Scores der *besten* Hyperparameter-Kombination für diesen Classifier
best_run_for_clf = clf_runs.loc[clf_runs['mean_test_score'].idxmax()]
scores_for_best_run = best_run_for_clf[fold_score_cols].values.flatten()
all_scores_data.append(scores_for_best_run)
classifier_order_for_boxplot.append(clf_name)
if all_scores_data: # Nur plotten, wenn Daten vorhanden sind
plt.figure(figsize=(10, 6))
plt.boxplot(all_scores_data, tick_labels=classifier_order_for_boxplot)
plt.xlabel("Classifier")
plt.ylabel(f"CV Scores ({search.scoring}) über die Folds (für die beste Parametereinstellung)")
plt.title("Verteilung der CV Scores für die beste Konfiguration jedes Classifiers")
plt.xticks(rotation=15, ha="right")
plt.tight_layout()
plt.show()
print("\nDetails zum besten gefundenen Modell insgesamt:")
print(f"Bester Estimator: {search.best_estimator_}")
print(f"Beste Parameter: {search.best_params_}")
print(f"Bester CV Score: {search.best_score_:.4f}")
# Evaluierung des besten Modells auf dem Test-Set
best_pipeline_final = search.best_estimator_
test_score_pipeline = best_pipeline_final.score(X_test, y_test)
print(f"\nTest-Set Genauigkeit des besten Modells (Pipeline-Ansatz): {test_score_pipeline:.4f}")Ausgabe:
Bester Classifier und Parameter: {'classifier': RandomForestClassifier(random_state=42), 'classifier__max_depth': 10, 'classifier__n_estimators': 50}
Bester Cross-Validation Score: 0.8963
Details zum besten gefundenen Modell insgesamt:
Bester Estimator: Pipeline(steps=[('classifier',
RandomForestClassifier(max_depth=10, n_estimators=50,
random_state=42))])
Beste Parameter: {'classifier': RandomForestClassifier(random_state=42), 'classifier__max_depth': 10, 'classifier__n_estimators': 50}
Bester CV Score: 0.8963
Test-Set Genauigkeit des besten Modells (Pipeline-Ansatz): 0.8850
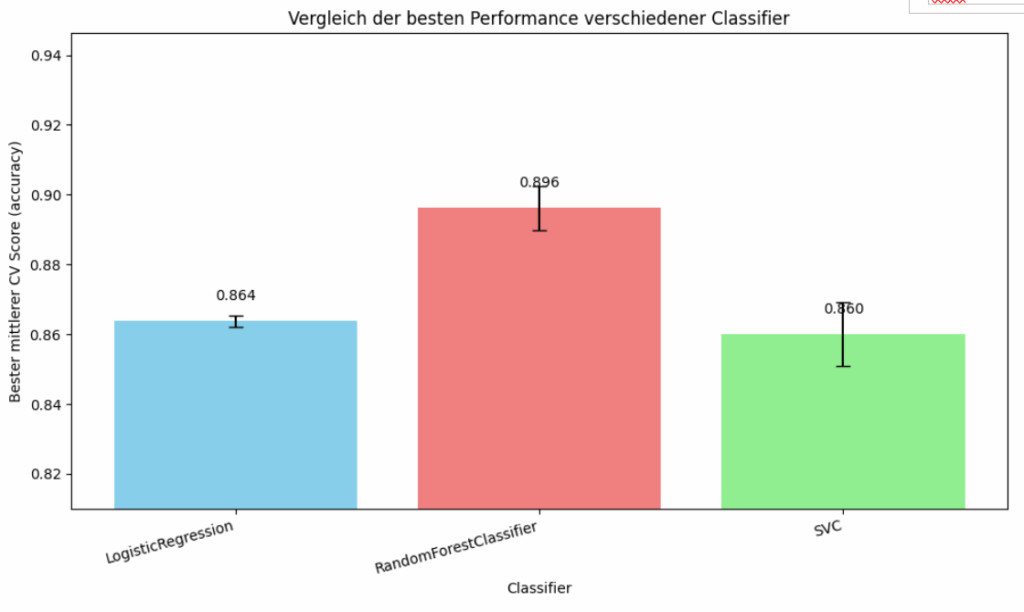
Das Balkendiagramm visualisiert den Leistungsvergleich der drei verschiedenen Klassifikationsalgorithmen: Logistic Regression, RandomForestClassifier und SVC. Die vertikale Achse stellt den besten mittleren Kreuzvalidierungs-Score dar, gemessen als Genauigkeit (Accuracy). Aus dem Diagramm geht hervor, dass der RandomForestClassifier mit einem Wert von etwa 0.896 die höchste mittlere Genauigkeit erzielt und somit in diesem Vergleich die beste Leistung zeigt. Darauf folgt die Logistic Regression mit circa 0.864 und der SVC mit einem knapp darunter liegenden Wert von ungefähr 0.860. Zusätzlich sind Fehlerbalken dargestellt, die die Standardabweichung oder Varianz der Leistung über die verschiedenen Durchläufe der Kreuzvalidierung für jede beste Parametereinstellung des jeweiligen Modells andeuten.
Insgesamt zeigt das Bild, dass der RandomForestClassifier unter den getesteten Bedingungen und basierend auf der mittleren Kreuzvalidierungsgenauigkeit die überlegene Leistung erbracht hat, während Logistic Regression und SVC eine vergleichbare, aber etwas geringere Performance aufweisen.
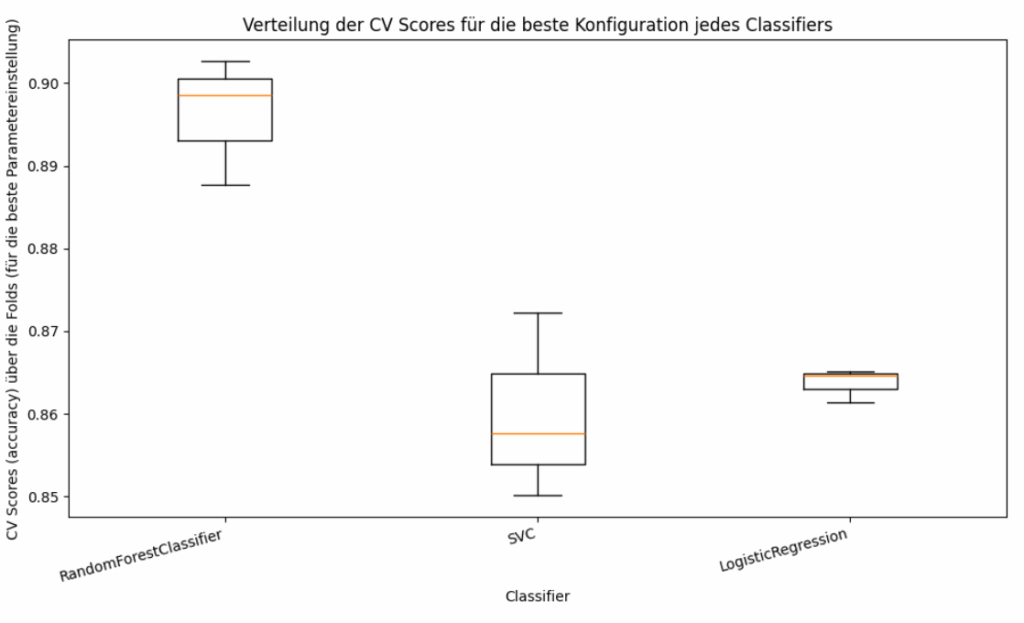
Das zweite Bild zeigt ein Boxplot-Diagramm, das die Verteilung der Kreuzvalidierungs-Scores (Genauigkeit) für die jeweils beste gefundene Konfiguration von drei verschiedenen Klassifikationsalgorithmen – RandomForestClassifier, SVC und LogisticRegression – darstellt. Die vertikale Achse bildet die CV-Genauigkeits-Scores ab, die über die verschiedenen Folds der Kreuzvalidierung für die beste Parametereinstellung jedes Modells erzielt wurden, während die horizontale Achse die Namen der Classifier auflistet. Für den RandomForestClassifier zeigt der Boxplot, dass die Scores tendenziell am höchsten liegen, mit einem Median (der orangen Linie in der Box) nahe 0.90. Die Box selbst, die die mittleren 50% der Scores repräsentiert, ist relativ kompakt, und die Whiskers, die die Streuung der meisten Datenpunkte anzeigen, erstrecken sich von etwa 0.887 bis knapp über 0.90, was auf eine generell hohe und einigermaßen konsistente Leistung über die Folds hinweist.
Im Gegensatz dazu weist der SVC einen niedrigeren Median-Score auf, der um 0.858 liegt. Die Box ist hier breiter, und die Whiskers haben eine größere Spannweite von circa 0.85 bis 0.873, was auf eine größere Variabilität oder Streuung der Leistung des SVC über die verschiedenen Kreuzvalidierungs-Folds für seine beste Konfiguration hindeutet.
Die LogisticRegression schließlich zeigt einen Median-Score um 0.864. Auffällig ist hier die sehr schmale Box und die kurzen Whiskers, was bedeutet, dass die Leistung der LogisticRegression mit ihrer besten Parametereinstellung über die Folds hinweg sehr konsistent ist, mit nur geringer Schwankung der Genauigkeitswerte.
Zusammenfassend illustriert dieses Diagramm vor allem die Stabilität und Streuung der Performance, wenn er mit seinen optimalen Hyperparametern auf verschiedenen Teilmengen der Trainingsdaten getestet wird. Es bestätigt, dass RandomForest die höchsten Scores erzielt, während Logistic Regression die konsistenteste Leistung zeigt, wenn auch auf einem etwas niedrigeren Niveau als RandomForest, und SVC sowohl niedrigere Scores als auch eine höhere Variabilität aufweist.
