TODO Für Tag 15
- SimpleImputer verstehen
- Beispiel zur Imputation in Daten
- SimpleImputer in einer Pipeline verwenden
SimpleImputer verstehen
Der SimpleImputer in scikit-learn ist ein Werkzeug zur Vorverarbeitung von Daten, das speziell dafür entwickelt wurde, fehlende Werte in numerischen oder kategorischen Datensätzen systematisch zu ersetzen. Er analysiert die vorhandenen Daten und füllt Lücken, die beispielsweise durch NaN-Werte entstanden sind, mithilfe einer vorher definierten Strategie. Dabei kann der Benutzer festlegen, ob fehlende Werte durch den arithmetischen Mittelwert, den Median, den häufigsten Wert oder einen konstanten Wert ersetzt werden sollen. Diese Strategien ermöglichen eine unkomplizierte und schnelle Bereinigung von unvollständigen Datensätzen, was besonders wichtig ist, da viele Machine-Learning-Algorithmen nicht in der Lage sind, mit fehlenden Werten umzugehen. Der Imputer wird zunächst mit den Trainingsdaten „gelernt“, wobei er die notwendigen Kennzahlen wie Mittelwert oder Modus berechnet, und kann anschließend auf neue Daten angewendet werden, um dort ebenfalls fehlende Werte konsistent zu ersetzen. Durch diese Vorgehensweise stellt der SimpleImputer sicher, dass der Umgang mit unvollständigen Daten reproduzierbar, nachvollziehbar und leicht in automatisierte Machine-Learning-Pipelines integrierbar ist.
Hier ein Beispiel, das zeigt, wie der SimpleImputer in scikit-learn mit verschiedenen Strategien (mean, median, most_frequent, constant) arbeitet. Das Beispiel vergleicht dabei, wie sich die verschiedenen Imputing-Strategien auf einen kleinen Datensatz mit fehlenden Werten auswirken.
import numpy as np
import matplotlib.pyplot as plt
from sklearn.impute import SimpleImputer
import pandas as pd
# Beispiel-Daten mit fehlenden Werten (NaN)
data = np.array([
[10, 2, np.nan],
[np.nan, 3, 9],
[8, np.nan, 7],
[9, 3, 6],
[np.nan, 4, np.nan]
])
# Verschiedene Strategien testen
strategies = ['mean', 'median', 'most_frequent', 'constant']
results = {}
for strategy in strategies:
if strategy == 'constant':
imputer = SimpleImputer(strategy=strategy, fill_value=0)
else:
imputer = SimpleImputer(strategy=strategy)
transformed = imputer.fit_transform(data)
results[strategy] = transformed
# Original-Daten als DataFrame (zur besseren Visualisierung)
df_original = pd.DataFrame(data, columns=["Feature 1", "Feature 2", "Feature 3"])
# Visualisierung mit Matplotlib
fig, axs = plt.subplots(1, len(strategies) + 1, figsize=(20, 5))
fig.suptitle("Vergleich verschiedener Imputing-Strategien mit SimpleImputer", fontsize=16)
# Originaldaten plotten
axs[0].imshow(df_original, cmap="viridis", interpolation="none")
axs[0].set_title("Original (mit NaN)")
axs[0].set_xticks([0, 1, 2])
axs[0].set_xticklabels(df_original.columns)
axs[0].set_yticks(range(len(df_original)))
axs[0].set_yticklabels([f"Sample {i+1}" for i in range(len(df_original))])
axs[0].tick_params(axis='x', rotation=45)
# Ergebnisse plotten
for i, strategy in enumerate(strategies):
axs[i+1].imshow(results[strategy], cmap="viridis", interpolation="none")
axs[i+1].set_title(strategy)
axs[i+1].set_xticks([0, 1, 2])
axs[i+1].set_xticklabels(df_original.columns)
axs[i+1].set_yticks(range(len(df_original)))
axs[i+1].set_yticklabels([f"Sample {i+1}" for i in range(len(df_original))])
axs[i+1].tick_params(axis='x', rotation=45)
plt.tight_layout(rect=[0, 0, 1, 0.95])
plt.show()
Der Datensatz enthält gezielt einige NaN-Werte. Für jede Strategie wird ein SimpleImputer konfiguriert und die Daten damit transformiert. Die Ergebnisse werden als Heatmaps (Farbmatrix) nebeneinander dargestellt, sodass man direkt sehen kann, wie sich die Imputation in den verschiedenen Features unterscheidet.
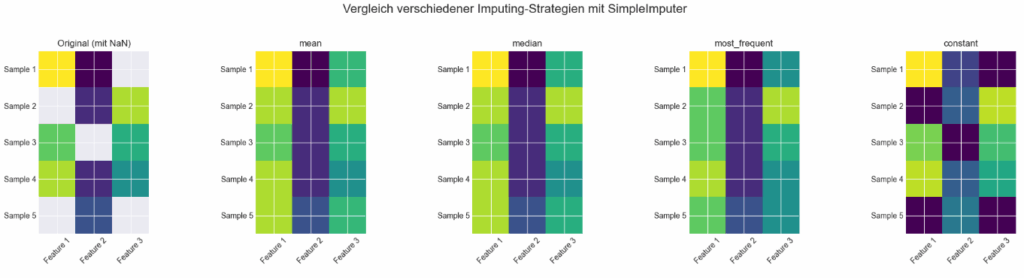
Die erste Heatmap zeigt die Originaldaten mit fehlenden Einträgen (dargestellt als helle Zellen). Die nachfolgenden vier Heatmaps illustrieren das Ergebnis der Datenvervollständigung unter Verwendung der Strategien „mean„, „median„, „most_frequent“ und „constant“ (hier mit dem Füllwert 0), wobei die fehlenden Stellen durch die berechneten oder zugewiesenen Werte ersetzt und farblich entsprechend den anderen Werten in der jeweiligen Spalte dargestellt sind, was die Auswirkungen jeder Methode auf den Datensatz visuell hervorhebt. Die Heatmaps zeigen jeweils fünf Stichproben (Sample 1 bis Sample 5) über drei Merkmale (Feature 1 bis Feature 3), wobei die Farbintensität die numerischen Werte repräsentiert.
Beispiel zur Imputation in Daten
Das Ziel der Imputation ist es, den Datensatz vollständig zu machen, damit er für weitere Analysen, Modellbildungen oder Visualisierungen verwendet werden kann, da viele statistische Verfahren und Algorithmen keine fehlenden Werte verarbeiten können. Statt einfach ganze Zeilen oder Spalten mit fehlenden Werten zu entfernen (was zu Informationsverlust führen kann), versucht die Imputation, die fehlenden Daten auf sinnvolle Weise zu ergänzen.
- Mittelwert-Imputation
Ersetzen durch den Durchschnittswert der jeweiligen Spalte. - Median-Imputation
Ersetzen durch den Medianwert der jeweiligen Spalte (weniger empfindlich gegenüber Ausreißern). - Most Frequent
Ersetzen durch den häufigsten Wert der jeweiligen Spalte (oft für kategoriale Daten verwendet, aber auch für numerische Daten möglich). - Konstante Imputation
Ersetzen durch einen festen, vordefinierten Wert (z. B. 0 oder einen Indikatorwert). - Komplexere Methoden
wie K-Nearest Neighbors (KNN) Imputation, statistische Modellierung (z. B. Regression) oder multiple Imputation, die versuchen, die fehlenden Werte basierend auf den vorhandenen Daten und deren Beziehungen zu schätzen.
Hier noch mal ein Beispiel, das zeigt, wie man fehlende Werte mit dem KNNImputer basierend auf den k-nächsten Nachbarn auffüllt. Das folgende Beispiel verwendet einen Beispiel-Datensatz mit NaN-Werten und stellen das Ergebnis des KNNImputers als Heatmap dar.
import numpy as np
import matplotlib.pyplot as plt
import pandas as pd
from sklearn.impute import KNNImputer
# Beispiel-Daten mit fehlenden Werten
data = np.array([
[10, 2, np.nan],
[np.nan, 3, 9],
[8, np.nan, 7],
[9, 3, 6],
[np.nan, 4, np.nan]
])
# DataFrame für bessere Achsenbeschriftung
df_original = pd.DataFrame(data, columns=["Feature 1", "Feature 2", "Feature 3"])
# KNNImputer konfigurieren (z. B. k=2)
knn_imputer = KNNImputer(n_neighbors=2, weights="uniform")
data_knn_imputed = knn_imputer.fit_transform(data)
df_imputed = pd.DataFrame(data_knn_imputed, columns=["Feature 1", "Feature 2", "Feature 3"])
# Plot: Heatmaps zum Vergleich
fig, axs = plt.subplots(1, 2, figsize=(12, 5))
fig.suptitle("KNN-Imputation von fehlenden Werten", fontsize=16)
# Originaldaten (mit NaN)
axs[0].imshow(df_original, cmap="viridis", interpolation="none")
axs[0].set_title("Originaldaten (mit NaN)")
axs[0].set_xticks([0, 1, 2])
axs[0].set_xticklabels(df_original.columns)
axs[0].set_yticks(range(len(df_original)))
axs[0].set_yticklabels([f"Sample {i+1}" for i in range(len(df_original))])
axs[0].tick_params(axis='x', rotation=45)
# Imputierte Daten
axs[1].imshow(df_imputed, cmap="viridis", interpolation="none")
axs[1].set_title("KNN-Imputation (k=2)")
axs[1].set_xticks([0, 1, 2])
axs[1].set_xticklabels(df_imputed.columns)
axs[1].set_yticks(range(len(df_imputed)))
axs[1].set_yticklabels([f"Sample {i+1}" for i in range(len(df_imputed))])
axs[1].tick_params(axis='x', rotation=45)
plt.tight_layout(rect=[0, 0, 1, 0.95])
plt.show()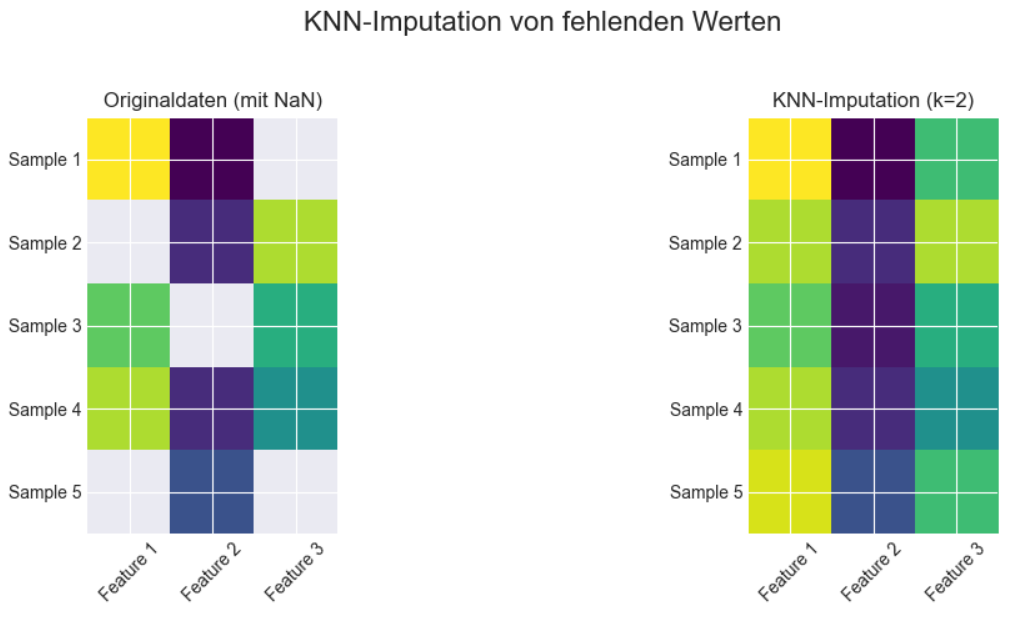
Der Plot zeigt einen Vergleich derselben Daten vor und nach der Anwendung der KNN-Imputation zur Behandlung fehlender Werte.
Die linke Heatmap „Originaldaten (mit NaN)“ stellt die ursprünglichen Daten dar. Sie ist als Matrix von 5 „Samples“ (Zeilen) und 3 „Features“ (Spalten) organisiert. Die Farbe jeder Zelle repräsentiert den Wert an dieser Position. Die Heatmap verdeutlicht die Verteilung der vorhandenen Datenpunkte und visualisiert klar die Positionen der fehlenden Einträge.
Die rechte Heatmap „KNN-Imputation (k=2)“ zeigt die gleichen Daten nachdem die fehlenden Werte durch KNN-Imputation ersetzt wurden. Der entscheidende Unterschied ist, dass keine Zellen mehr leer oder in der NaN-Farbe dargestellt sind. Die zuvor fehlenden Zellen wurden mit Werten gefüllt. Diese neuen Werte wurden mithilfe des KNN-Imputers berechnet, wobei n_neighbors=2 eingestellt ist. Das bedeutet, dass für jede fehlende Zelle der Algorithmus die 2 „nächsten“ Datenpunkte in den Daten sucht (basierend auf den Features, die sowohl im aktuellen Eintrag als auch im potentiellen Nachbarn vorhanden sind) und den fehlenden Wert dann durch den Durchschnitt der Werte in der betreffenden Feature von diesen 2 Nachbarn ersetzt. Man sieht deutlich, dass die imputierten Werte eine Farbe haben, die in das bestehende Farbspektrum der jeweiligen Spalte/des jeweiligen Bereichs passt, was darauf hindeutet, dass die Imputation Werte liefert, die plausibel im Kontext der umgebenden Daten liegen.
SimpleImputer in einer Pipeline verwenden
Man kann den SimpleImputer auch in einer Pipeline verwenden. Das folgenden Beispiel, zeigt wie man den SimpleImputer innerhalb einer Pipeline verwendet.
import numpy as np
import pandas as pd
from sklearn.model_selection import train_test_split
from sklearn.compose import ColumnTransformer
from sklearn.pipeline import Pipeline
from sklearn.impute import SimpleImputer
from sklearn.preprocessing import StandardScaler, OneHotEncoder
from sklearn.linear_model import LogisticRegression
from sklearn.metrics import classification_report, accuracy_score
# Synthetische Daten mit fehlenden Werten generieren
# Anzahl der Samples
n_samples = 200
# Numerische Features (mit NaNs)
np.random.seed(42) # für Reproduzierbarkeit
data_num = np.random.rand(n_samples, 3) * 100
# Füge NaNs zufällig hinzu
nan_indices_num = np.random.choice(n_samples * 3, size=int(n_samples * 3 * 0.1), replace=False) # 10% NaNs
data_num.ravel()[nan_indices_num] = np.nan
# Kategorische Features (mit NaNs)
categories_cat1 = ['A', 'B', 'C', 'D']
categories_cat2 = ['X', 'Y']
data_cat1 = np.random.choice(categories_cat1, size=(n_samples, 1))
data_cat2 = np.random.choice(categories_cat2, size=(n_samples, 1))
data_cat = np.hstack((data_cat1, data_cat2))
# Füge NaNs zufällig hinzu
nan_indices_cat = np.random.choice(n_samples * 2, size=int(n_samples * 2 * 0.15), replace=False) # 15% NaNs
data_cat.ravel()[nan_indices_cat] = np.nan
# Konvertiere zu String, damit NaN einheitlich behandelt wird
data_cat = data_cat.astype(object) # Wichtig für NaN in Objekt-Arrays
for idx in nan_indices_cat:
data_cat.ravel()[idx] = np.nan
# Target Variable (abhängig von Features, für ein sinnvolles Modell)
# Einfaches Beispiel: Target ist 1, wenn num_feature_0 > 50 ODER cat_feature_0 ist 'A'
# Muss vor dem Hinzufügen von NaNs erzeugt werden oder NaNs handhaben
# Für dieses Beispiel erstellen wir es einfach basierend auf den ursprünglichen, nicht-NaN Werten
target = ((data_num[:, 0] > 50) | (data_cat1.flatten() == 'A')).astype(int)
# Kombiniere alles in einem Pandas DataFrame
feature_names = ['num_feature_0', 'num_feature_1', 'num_feature_2', 'cat_feature_0', 'cat_feature_1']
X = pd.DataFrame(np.hstack((data_num, data_cat)), columns=feature_names)
y = pd.Series(target, name='target')
print("Beispieldaten mit NaNs:")
print(X.head())
print("\nAnzahl NaNs pro Spalte:")
print(X.isnull().sum())
print("-" * 30)
# Daten in Trainings- und Testsets aufteilen
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.2, random_state=42)
print(f"Trainingsdaten Größe: {X_train.shape}")
print(f"Testdaten Größe: {X_test.shape}")
print("-" * 30)
# Pipeline für die Vorverarbeitung erstellen
# Definieren der Spalten nach Datentyp
numerical_features = ['num_feature_0', 'num_feature_1', 'num_feature_2']
categorical_features = ['cat_feature_0', 'cat_feature_1']
# Erstelle separate Pipelines für numerische und kategorische Features
# Pipeline für numerische Features: Imputation (Mittelwert) -> Skalierung
numerical_transformer = Pipeline(steps=[
('imputer', SimpleImputer(strategy='mean')), # Füllt NaNs mit Spaltenmittelwert
('scaler', StandardScaler()) # Skaliert die Features auf Mittelwert 0, Varianz 1
])
# Pipeline für kategorische Features: Imputation (häufigster Wert) -> One-Hot-Encoding
categorical_transformer = Pipeline(steps=[
('imputer', SimpleImputer(strategy='most_frequent')), # Füllt NaNs mit dem häufigsten Wert
('onehot', OneHotEncoder(handle_unknown='ignore')) # Konvertiert Kategorien in numerische Vektoren
])
# Kombiniere die Pipelines für verschiedene Spalten mit ColumnTransformer
preprocessor = ColumnTransformer(
transformers=[
('num', numerical_transformer, numerical_features),
('cat', categorical_transformer, categorical_features)
])
print("Vorverarbeitungs-Pipeline erstellt.")
print("-" * 30)
# Die gesamte Pipeline erstellen (Vorverarbeitung + Modell)
# Füge das Modell zur Pipeline hinzu
model = LogisticRegression(random_state=42)
full_pipeline = Pipeline(steps=[('preprocessor', preprocessor),
('classifier', model)])
print("Gesamte Pipeline (Vorverarbeitung + Modell) erstellt.")
print(full_pipeline)
print("-" * 30)
# Pipeline trainieren
# Trainiere die gesamte Pipeline auf den Trainingsdaten
# Während fit() werden ausgeführt:
# - preprocessor.fit_transform(X_train)
# - num_pipeline.fit_transform() für num_features
# - cat_pipeline.fit_transform() für cat_features
# - classifier.fit(transformed_X_train, y_train)
print("Trainiere die Pipeline...")
full_pipeline.fit(X_train, y_train)
print("Pipeline-Training abgeschlossen.")
print("-" * 30)
# Modell auf den Testdaten evaluieren
# Mache Vorhersagen auf den Testdaten
# Während predict() werden ausgeführt:
# - preprocessor.transform(X_test) (fit wurde auf X_train gemacht, hier nur transform)
# - classifier.predict(transformed_X_test)
y_pred = full_pipeline.predict(X_test)
# Bewerte die Performance
accuracy = accuracy_score(y_test, y_pred)
report = classification_report(y_test, y_pred)
print(f"Modell-Genauigkeit auf Testdaten: {accuracy:.4f}")
print("\nKlassifikationsbericht auf Testdaten:")
print(report)
print("-" * 30)
# Optional: Manuelle Überprüfung der Imputation
# Dies ist normalerweise nicht nötig, da die Pipeline alles intern macht
# Transformation nur auf den Trainingsdaten anwenden (um Ergebnisse zu sehen)
# X_train_processed = preprocessor.fit_transform(X_train) # Nicht tun, da fit() bereits in der Pipeline auf X_train lief
# Stattdessen nur transform() nach dem fit() der Pipeline nutzen
X_train_processed = full_pipeline.named_steps['preprocessor'].transform(X_train)
X_test_processed = full_pipeline.named_steps['preprocessor'].transform(X_test)
print("\nBeispiel der vorverarbeiteten Trainingsdaten (erste 5 Samples):")
# Da OneHotEncoder verwendet wird, sind die Spaltennamen nicht trivial
# Man kann die Feature-Namen aus dem OneHotEncoder extrahieren
# onehot_features = full_pipeline.named_steps['preprocessor'].named_transformers_['cat'].named_steps['onehot'].get_feature_names_out(categorical_features)
# processed_feature_names = numerical_features + list(onehot_features)
# df_processed = pd.DataFrame(X_train_processed, columns=processed_feature_names)
print(X_train_processed[:5])
print("\nNaNs in den vorverarbeiteten Daten (sollten keine mehr da sein):")
print(np.isnan(X_train_processed).sum())Das Beispiel generiert synthetische Daten mit mehreren numerischen und kategorischen Spalten mit fehlende Werte (NaNs) bei beiden Datentypen. Dann verwendet es den ColumnTransformer, um verschiedene Vorverarbeitungsschritte auf unterschiedliche Spalten anzuwenden:
- Numerische Spalten
Imputation mit dem Mittelwert (SimpleImputer), gefolgt von Skalierung (StandardScaler). - Kategorische Spalten
Imputation mit dem häufigsten Wert (SimpleImputer), gefolgt von One-Hot-Encoding (OneHotEncoder).
Die Ausgabe des Codes sieht dann wie folgt aus:
Beispieldaten mit NaNs:
num_feature_0 num_feature_1 num_feature_2 cat_feature_0 cat_feature_1
0 37.454012 95.071431 73.199394 B Y
1 59.865848 15.601864 15.599452 C Y
2 5.808361 86.617615 60.111501 D Y
3 70.807258 2.058449 96.990985 C Y
4 83.244264 21.233911 18.182497 D X
Anzahl NaNs pro Spalte:
num_feature_0 22
num_feature_1 19
num_feature_2 19
cat_feature_0 32
cat_feature_1 28
dtype: int64
------------------------------
Trainingsdaten Größe: (160, 5)
Testdaten Größe: (40, 5)
------------------------------
Vorverarbeitungs-Pipeline erstellt.
------------------------------
Gesamte Pipeline (Vorverarbeitung + Modell) erstellt.
Pipeline(steps=[('preprocessor',
ColumnTransformer(transformers=[('num',
Pipeline(steps=[('imputer',
SimpleImputer()),
('scaler',
StandardScaler())]),
['num_feature_0',
'num_feature_1',
'num_feature_2']),
('cat',
Pipeline(steps=[('imputer',
SimpleImputer(strategy='most_frequent')),
('onehot',
OneHotEncoder(handle_unknown='ignore'))]),
['cat_feature_0',
'cat_feature_1'])])),
('classifier', LogisticRegression(random_state=42))])
------------------------------
Trainiere die Pipeline...
Pipeline-Training abgeschlossen.
------------------------------
Modell-Genauigkeit auf Testdaten: 0.9000
Klassifikationsbericht auf Testdaten:
precision recall f1-score support
0 1.00 0.71 0.83 14
1 0.87 1.00 0.93 26
accuracy 0.90 40
macro avg 0.93 0.86 0.88 40
weighted avg 0.91 0.90 0.90 40
------------------------------
Beispiel der vorverarbeiteten Trainingsdaten (erste 5 Samples):
[[ 5.23759727e-16 5.18541574e-01 -1.05644291e+00 0.00000000e+00
0.00000000e+00 0.00000000e+00 1.00000000e+00 0.00000000e+00
1.00000000e+00]
[ 4.68963889e-01 -1.12214318e+00 -4.59455959e-01 1.00000000e+00
0.00000000e+00 0.00000000e+00 0.00000000e+00 1.00000000e+00
0.00000000e+00]
[ 4.41428580e-01 2.51465967e-16 1.16268686e+00 0.00000000e+00
0.00000000e+00 1.00000000e+00 0.00000000e+00 0.00000000e+00
1.00000000e+00]
[-1.87271172e+00 1.12014482e+00 8.19792360e-01 1.00000000e+00
0.00000000e+00 0.00000000e+00 0.00000000e+00 0.00000000e+00
1.00000000e+00]
[ 1.64952994e+00 1.43826893e+00 -9.90281228e-01 0.00000000e+00
1.00000000e+00 0.00000000e+00 0.00000000e+00 1.00000000e+00
0.00000000e+00]]
NaNs in den vorverarbeiteten Daten (sollten keine mehr da sein):
0
Das Beispiel zeigt eine Pipeline, die sicherstellt, dass die Imputation und Skalierung/Kodierung konsistent auf Trainings- und Testdaten angewendet wird. Die Parameter (Mittelwerte, etc.) werden aus den Trainingsdaten gelernt, was eine realistische Bewertung der Modellleistung ermöglicht. Das Beispiel zeigt, wie man die Imputation auf einfache Art und Weise in eine Pipeline integriert.
