TODO für Tag 8
- Implementierung von DecisionTreeClassifier
- Visualisierung des Baumes (z.B. mit export_graphviz / plot_tree)
Der DecisionTreeClassifier
Der DecisionTreeClassifier aus scikit-learn ist ein einfacher, schneller und interpretierbarer Klassifikator, der auf Entscheidungsbäumen basiert. Er lernt Entscheidungsregeln, indem er die Trainingsdaten rekursiv nach den „besten“ Attributen aufspaltet, um die Klassen möglichst sauber zu trennen.
Hier ein einfaches Beispiel, bei dem der Iris-Datensatz mit dem DecisionTreeClassifier klassifiziert wird:
from sklearn.datasets import load_iris
from sklearn.tree import DecisionTreeClassifier
from sklearn.model_selection import train_test_split
from sklearn.metrics import accuracy_score
# 1. Daten laden
X, y = load_iris(return_X_y=True)
# 2. Trainings- und Testdaten aufteilen
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.3, random_state=42)
# 3. Modell erstellen (mit Begrenzung der Tiefe, um Overfitting zu vermeiden)
clf = DecisionTreeClassifier(max_depth=3, random_state=42)
# 4. Trainieren
clf.fit(X_train, y_train)
# 5. Vorhersagen
y_pred = clf.predict(X_test)
# 6. Bewertung
accuracy = accuracy_score(y_test, y_pred)
print(f"Genauigkeit: {accuracy:.2f}")
# Optional: Baum visualisieren (wenn du willst)
from sklearn.tree import plot_tree
import matplotlib.pyplot as plt
plt.figure(figsize=(12,8))
plot_tree(clf, filled=True, feature_names=load_iris().feature_names, class_names=load_iris().target_names)
plt.show()Mit dem Code erhält man eine Genauigkeit von 100% und das beste darin ist, dass man mit plot_tree auch noch den Entscheidungsbaum visualisieren kann.
Visualisierung des Baumes mit plot_tree
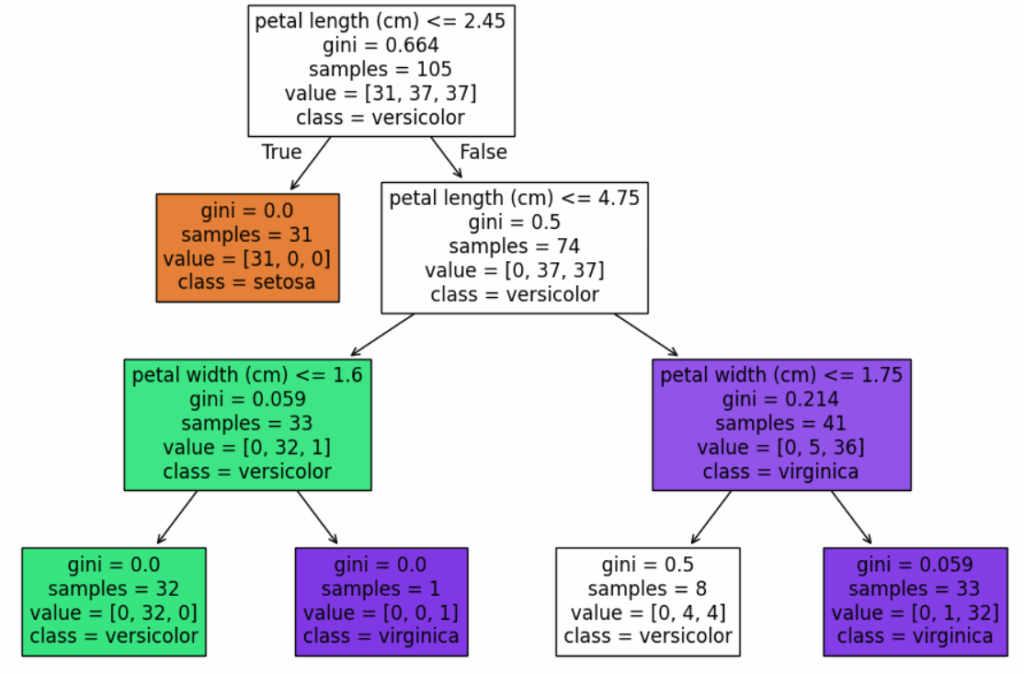
Der Baum zeigt genau auf, wie das Modell zu einer Klassifikation eines Datensatzes kommt.
- Wurzelknoten (ganz oben):
- Spalte:
petal length (cm) - Grenze:
<= 2.45 - Bedeutung: Wenn die Blütenblatt-Länge kleiner oder gleich 2.45 cm ist → links, sonst → rechts.
- Gini = 0.664: Hohe Unreinheit, also eine Mischung von Klassen.
- Samples = 105: Anzahl der Trainingsdaten an diesem Punkt.
- Spalte:
Linker Teil (True, also petal length <= 2.45):
- Gini = 0.0 → perfekte Reinheit (nur eine Klasse).
- Samples = 31 → alle diese Samples sind setosa (orange Box).
- Alle Samples mit kurzem Blütenblatt (
<= 2.45) sind safe setosa → fertig, keine weiteren Splits nötig.
Rechter Teil (False, also petal length > 2.45):
- Neuer Split bei
petal length <= 4.75 - Gini = 0.5: Immer noch gemischt, aber besser als ganz oben.
- Samples = 74
- Baum teilt sich wieder:
Linker Zweig (petal length <= 4.75):
- Split auf
petal width <= 1.6 - Sehr niedrige Gini-Werte, also fast perfekt getrennt:
- Links (
petal width <= 1.6): fast alles versicolor.- 32 versicolor, 0 setosa, 0 virginica.
- Rechts: ein einzelnes Sample → virginica.
- Links (
- Praktisch perfekte Trennung.
Rechter Zweig (petal length > 4.75):
- Split auf
petal width <= 1.75 - Samples = 41
- Links (
petal width <= 1.75): bisschen gemischt (4 versicolor, 4 virginica). - Rechts: fast alles virginica (32 virginica, 1 versicolor).
- Links (
Der dargestellte Entscheidungsbaum klassifiziert den Iris-Datensatz sehr effizient und erreicht eine nahezu perfekte Trennung der drei Klassen mit nur wenigen Splits und einer überschaubaren Baumtiefe. Besonders auffällig ist, dass einfache Schwellenwerte für Blütenblattlänge und -breite ausreichen, um die meisten Beispiele sauber zu trennen. Die Gini-Indizes zeigen, dass der Baum sehr schnell klare Entscheidungen trifft und die Trainingsdaten sauber segmentiert.
