TODO für Tag 18
- Cross-Validation innerhalb einer Pipeline durchführen
Cross-Validation innerhalb einer Pipeline durchführen
Die Durchführung von Kreuzvalidierung innerhalb einer Pipeline ist ein Ansatz, der dabei hilft, robuste und verlässliche Modelle zu entwickeln. Das Prinzip ist, sicherzustellen, dass alle Schritte der Datenvorverarbeitung, wie Skalierung, Imputation fehlender Werte oder Merkmalsextraktion, korrekt innerhalb jedes einzelnen Kreuzvalidierungs-Splits angewendet werden. Dies verhindert ein sogenanntes „Data Leakage“, bei dem Informationen aus den Testdaten (oder Validierungsdaten innerhalb eines Folds) unbeabsichtigt in den Trainingsprozess der Vorverarbeitungsschritte einfließen und so zu optimistischen und unrealistischen Leistungsschätzungen führen.
Eine Pipeline führt eine Sequenz von Datentransformatoren und einem finalen Schätzer (dem eigentlichen Modell) zu einem einzigen Objekt zusammen. Wenn dieses Pipeline-Objekt dann einer Kreuzvalidierungsfunktion wie GridSearchCV übergeben wird, behandelt die Kreuzvalidierungslogik die gesamte Pipeline als eine Einheit.
Die Daten werden zunächst in einen Trainings- und einen Validierungssatz für diesen spezifischen Fold aufgeteilt. Anschließend wird die Pipeline auf die Trainingsdaten dieses Folds „angepasst“. Dabei werden nacheinander alle Transformatoren in der Pipeline auf die Trainingsdaten des aktuellen Folds angewendet. Jeder Transformator lernt seine Parameter ausschließlich aus diesen Trainingsdaten. Nachdem die Trainingsdaten transformiert wurden, wird der finale Schätzer am Ende der Pipeline ebenfalls nur mit diesen transformierten Trainingsdaten des aktuellen Folds trainiert.
Sobald die Pipeline auf den Trainingsdaten des Folds angepasst wurde, wird sie verwendet, um Vorhersagen für die Validierungsdaten desselben Folds zu treffen. Wichtig ist hierbei, dass die auf den Trainingsdaten gelernten Parameter der Transformatoren nun verwendet werden, um die Validierungsdaten zu transformieren. Es findet also kein erneutes Anpassen der Transformatoren auf den Validierungsdaten statt, sondern lediglich eine Anwendung der bereits gelernten Transformation. Schließlich wird die Leistung des Schätzers auf diesen transformierten Validierungsdaten bewertet.
Dieser Vorgang wird für jeden einzelnen Fold der Kreuzvalidierung wiederholt, wobei jeder Fold einmal als Validierungssatz dient und die restlichen Folds als Trainingssatz. Die Pipeline wird also in jedem Fold neu auf den jeweiligen Trainingsdaten angepasst. Am Ende erhält man eine Reihe von Leistungswerten, einen für jeden Fold, die dann aggregiert werden können, um eine robuste Schätzung der Modellgüte zu erhalten.
Der entscheidende Vorteil ist, dass die Vorverarbeitungsschritte in jedem Fold der Kreuzvalidierung strikt getrennt und unabhängig voneinander auf den jeweiligen Trainingsdaten gelernt werden. Dies simuliert realistisch, wie das Modell auf ungesehenen Daten performen würde, da auch hier die Vorverarbeitungsparameter aus den Trainingsdaten abgeleitet und dann auf die neuen Daten angewendet werden müssten. Die Verwendung von Pipelines in Verbindung mit Kreuzvalidierung ist somit ein wichtiges Werkzeugt für methodisch sauberes und zuverlässiges maschinelles Lernen.
Beispiel:
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
from sklearn.datasets import fetch_covtype # Größerer Datensatz
from sklearn.model_selection import train_test_split, GridSearchCV
from sklearn.preprocessing import StandardScaler
from sklearn.svm import SVC
from sklearn.ensemble import RandomForestClassifier
from sklearn.linear_model import LogisticRegression
from sklearn.pipeline import Pipeline
from sklearn.metrics import accuracy_score, classification_report
# Daten laden
print("Lade Covertype-Datensatz...")
# return_X_y=True gibt direkt NumPy-Arrays zurück
# as_frame=False ist der Standard, um NumPy-Arrays zu bekommen
X_all, y_all = fetch_covtype(return_X_y=True)
print(f"Originale Datensatzgröße: X={X_all.shape}, y={y_all.shape}")
# Für eine schnelle Demonstration werden nur die ersten 30000 Samples
# verwendet, da GridSearchCV sonst sehr langsam ist
# Für echte Projekte sollte man mehr Daten nutzen.
n_samples_to_use = 30000
X_subset, y_subset = X_all[:n_samples_to_use], y_all[:n_samples_to_use]
print(f"Datensatzgröße für das Beispiel: X={X_subset.shape}, y={y_subset.shape}")
print(f"Klassen: {np.unique(y_subset)}")
# Aufteilen in Trainings- und Testdaten
X_train, X_test, y_train, y_test = train_test_split(
X_subset, y_subset, test_size=0.3, random_state=42, stratify=y_subset
)
print(f"Größe der Trainingsdaten: {X_train.shape}")
print(f"Größe der Testdaten: {X_test.shape}")
# Pipeline-Grundgerüst definieren
# Der 'classifier'-Schritt ist ein Platzhalter, der durch GridSearchCV mit
# den tatsächlichen Modellen (SVC, RandomForest, LogisticRegression) ersetzt wird.
base_pipeline = Pipeline([
('scaler', StandardScaler()), # Schritt 1: Daten skalieren
('classifier', SVC()) # Platzhalter-Klassifikator
])
# Parameter-Grid für GridSearchCV definieren
# Dies wird eine LISTE von Dictionaries. Jedes Dictionary definiert einen
# zu testenden Estimator und seine Parameter.
param_grid = [
{
'classifier': [SVC(random_state=42)],
'classifier__C': [0.1, 1, 10],
'classifier__gamma': [0.01, 0.1, 'scale'], # 'scale' ist ein guter Standard
'classifier__kernel': ['rbf'] # Für SVC oft am besten, linear wäre auch eine Option
},
{
'classifier': [RandomForestClassifier(random_state=42)],
'classifier__n_estimators': [50, 100, 150], # Weniger Bäume für schnellere Ausführung
'classifier__max_depth': [5, 10, None],
'classifier__min_samples_split': [2, 5]
},
{
'classifier': [LogisticRegression(random_state=42, solver='liblinear', max_iter=200)], # liblinear gut für kleinere Datensätze und L1/L2
'classifier__C': [0.01, 0.1, 1, 10],
'classifier__penalty': ['l1', 'l2']
}
]
# GridSearchCV instanziieren
# cv=3 für schnellere Ausführung. Für robustere Ergebnisse
# ist cv=5 oder cv=10 besser.
grid_search = GridSearchCV(
estimator=base_pipeline,
param_grid=param_grid,
cv=3, # Kreuzvalidierungs-Folds (weniger für schnellere Ausführung)
scoring='accuracy',
verbose=2, # Mehr Output
n_jobs=-1, # Alle CPU-Kerne nutzen
refit=True # Das beste Modell wird am Ende auf den gesamten Trainingsdaten neu trainiert
)
# GridSearchCV auf den Trainingsdaten fitten
print("\nStarte GridSearchCV (dies kann einige Zeit dauern)...")
grid_search.fit(X_train, y_train)
# Ergebnisse von GridSearchCV anzeigen
print("\nBeste gefundene Parameterkombination:")
print(grid_search.best_params_)
print(f"\nBeste Cross-Validation Accuracy auf den Trainingsdaten: {grid_search.best_score_:.4f}")
best_pipeline_overall = grid_search.best_estimator_
print("\nBestes Modell (Pipeline) insgesamt:")
print(best_pipeline_overall)
# Das beste Modell auf den separaten Testdaten evaluieren
y_pred_test = best_pipeline_overall.predict(X_test)
test_accuracy = accuracy_score(y_test, y_pred_test)
print(f"\nAccuracy des besten Modells auf den ungesehenen Testdaten: {test_accuracy:.4f}")
print("\nClassification Report auf den Testdaten:")
print(classification_report(y_test, y_pred_test, zero_division=0))
# Ergebnisse visualisieren
# Mittlere CV-Accuracy für jede getestete Parameterkombination.
# Top-Ergebnisse auflisten oder die Performance
# der besten Konfiguration jedes Modelltyps vergleichen.
results_df = pd.DataFrame(grid_search.cv_results_)
# Relevante Spalten auswählen und nach bestem Score sortieren
# 'param_classifier' zeigt, welches Modell verwendet wurde
results_df_sorted = results_df.sort_values(by='rank_test_score')
print("\nTop 5 Konfigurationen laut GridSearchCV:")
print(results_df_sorted[['param_classifier', 'mean_test_score', 'std_test_score', 'params']].head())
# Visualisierung der besten mittleren CV-Scores pro Modelltyp
# Extrahieren des Modellnamens für eine übersichtlichere Darstellung
def get_model_name(model_obj_str):
return str(model_obj_str).split('(')[0]
results_df_sorted['model_type'] = results_df_sorted['param_classifier'].apply(get_model_name)
# Finde den besten Score für jeden Modelltyp
best_score_per_model_type = results_df_sorted.loc[results_df_sorted.groupby('model_type')['mean_test_score'].idxmax()]
plt.figure(figsize=(12, 7))
bars = plt.bar(best_score_per_model_type['model_type'], best_score_per_model_type['mean_test_score'], color=['skyblue', 'lightgreen', 'salmon'])
plt.xlabel("Modelltyp")
plt.ylabel("Beste mittlere Cross-Validation Accuracy")
plt.title("Vergleich der besten CV-Accuracy pro Modelltyp")
plt.ylim(0, 1) # Accuracy ist zwischen 0 und 1
# Werte auf den Balken anzeigen
for bar in bars:
yval = bar.get_height()
plt.text(bar.get_x() + bar.get_width()/2.0, yval + 0.01, f'{yval:.4f}', ha='center', va='bottom')
plt.xticks(rotation=15)
plt.tight_layout()
plt.show()
# Optional: Heatmaps für spezifische Modelle, falls gewünscht und Parameter passen
# Beispiel für SVC (wenn es unter den Top-Modellen ist und man C vs Gamma plotten will)
# Dies erfordert eine Filterung der `results_df` für den spezifischen Modelltyp und
# ein Reshaping der Scores, ähnlich wie im vorherigen Beispiel.
# Da die Parameter-Grids unterschiedlich sind, müsste dies für jedes Modell angepasst werden.
# Hier ein Beispiel, wie man für SVC vorgehen könnte, wenn man es detaillierter analysieren möchte:
svc_results = results_df[results_df['param_classifier'].astype(str).str.contains('SVC')].copy() # .copy() um SettingWithCopyWarning zu vermeiden
if not svc_results.empty:
# Sicherstellen, dass die Parameter auch als Spalten existieren
if 'param_classifier__C' in svc_results.columns and 'param_classifier__gamma' in svc_results.columns:
# Konvertiere Parametertypen für korrekte Pivotierung
svc_results['param_classifier__C'] = svc_results['param_classifier__C'].astype(float)
# Gamma kann 'scale' sein, was nicht direkt numerisch ist. Muss ggf. behandelt werden.
# Für eine einfache Heatmap filtern wir auf numerische Gammas oder ersetzen 'scale'.
# Nur numerische Gammas im Grid für SVC annehmen.
# Für dieses Beispiel wird 'scale' einfach ausgefiltert, falls es Probleme macht.
svc_results_numeric_gamma = svc_results[pd.to_numeric(svc_results['param_classifier__gamma'], errors='coerce').notnull()].copy()
if not svc_results_numeric_gamma.empty:
svc_results_numeric_gamma['param_classifier__gamma'] = svc_results_numeric_gamma['param_classifier__gamma'].astype(float)
try:
svc_pivot = svc_results_numeric_gamma.pivot_table(
index='param_classifier__C',
columns='param_classifier__gamma',
values='mean_test_score'
)
plt.figure(figsize=(8, 6))
plt.imshow(svc_pivot, interpolation='nearest', cmap=plt.cm.viridis, aspect='auto')
plt.xlabel('gamma')
plt.ylabel('C')
plt.colorbar(label='Mean CV Accuracy (SVC)')
plt.xticks(np.arange(len(svc_pivot.columns)), svc_pivot.columns)
plt.yticks(np.arange(len(svc_pivot.index)), svc_pivot.index)
plt.title('GridSearchCV Scores für SVC (C vs Gamma)')
plt.tight_layout()
plt.show()
except Exception as e:
print(f"\nKonnte keine SVC Heatmap erstellen: {e}")
print("Möglicherweise waren nicht genügend numerische Gamma-Werte für eine Pivot-Tabelle vorhanden oder die Form passte nicht.")
else:
print("\nKeine SVC-Ergebnisse mit rein numerischen Gamma-Werten für Heatmap gefunden.")
else:
print("\nSVC Parameter für Heatmap (C, gamma) nicht in Ergebnissen gefunden.")
else:
print("\nKeine SVC-Ergebnisse in GridSearchCV-Resultaten gefunden für detaillierte Heatmap.")
Ausgabe:
Lade Covertype-Datensatz...
Originale Datensatzgröße: X=(581012, 54), y=(581012,)
Verwendete Datensatzgröße für das Beispiel: X=(30000, 54), y=(30000,)
Einzigartige Klassen: [1 2 3 4 5 6 7]
Größe der Trainingsdaten: (21000, 54)
Größe der Testdaten: (9000, 54)
Starte GridSearchCV (dies kann einige Zeit dauern)...
Fitting 3 folds for each of 35 candidates, totalling 105 fits
Beste gefundene Parameterkombination:
{'classifier': RandomForestClassifier(random_state=42), 'classifier__max_depth': None, 'classifier__min_samples_split': 2, 'classifier__n_estimators': 150}
Beste Cross-Validation Accuracy auf den Trainingsdaten: 0.8967
Bestes Modell (Pipeline) insgesamt:
Pipeline(steps=[('scaler', StandardScaler()),
('classifier',
RandomForestClassifier(n_estimators=150, random_state=42))])
Accuracy des besten Modells auf den ungesehenen Testdaten: 0.9079
Classification Report auf den Testdaten:
precision recall f1-score support
1 0.88 0.81 0.84 1431
2 0.94 0.94 0.94 4258
3 0.83 0.80 0.82 648
4 0.91 0.97 0.94 648
5 0.90 0.92 0.91 719
6 0.84 0.86 0.85 648
7 0.94 0.98 0.96 648
accuracy 0.91 9000
macro avg 0.89 0.90 0.89 9000
weighted avg 0.91 0.91 0.91 9000
Top 5 Konfigurationen laut GridSearchCV:
param_classifier mean_test_score std_test_score \
23 RandomForestClassifier(random_state=42) 0.896667 0.000992
22 RandomForestClassifier(random_state=42) 0.895571 0.001050
26 RandomForestClassifier(random_state=42) 0.892476 0.001342
21 RandomForestClassifier(random_state=42) 0.892429 0.000808
25 RandomForestClassifier(random_state=42) 0.891238 0.001477
params
23 {'classifier': RandomForestClassifier(random_s...
22 {'classifier': RandomForestClassifier(random_s...
26 {'classifier': RandomForestClassifier(random_s...
21 {'classifier': RandomForestClassifier(random_s...
25 {'classifier': RandomForestClassifier(random_s...
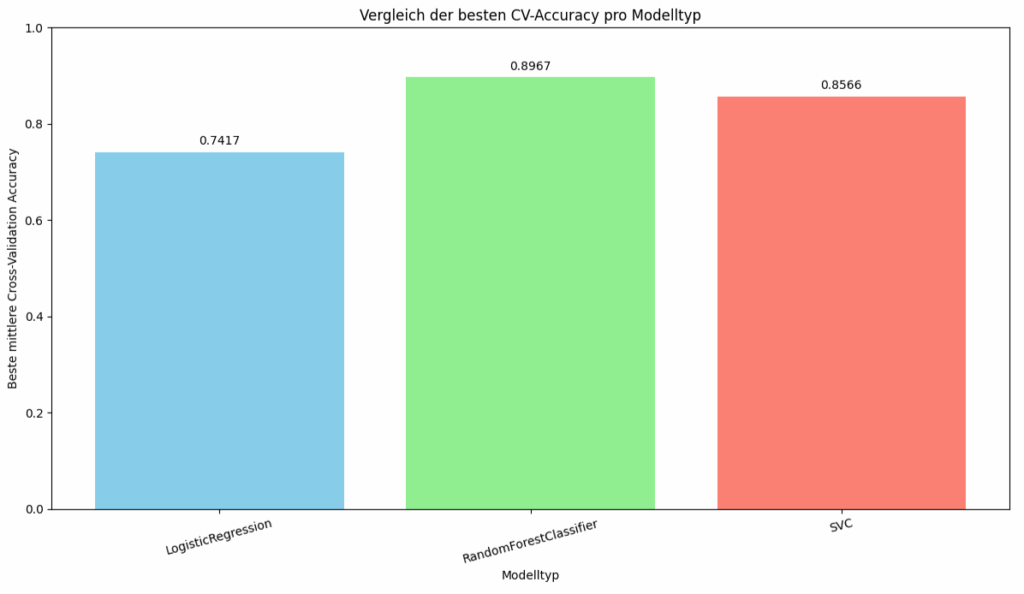
Das Balkendiagramm stellt die Leistung der drei verschiedenen Modell dar, gemessen an ihrer besten mittleren Kreuzvalidierungs-Genauigkeit (engl. Cross-Validation Accuracy). Die LogisticRegression erreicht eine beste mittlere Kreuzvalidierungs-Genauigkeit von etwa 74.2%. Der RandomForestClassifier zeigt die höchste Leistung mit einer besten mittleren Kreuzvalidierungs-Genauigkeit von ungefähr 89.7%. Der SVC (Support Vector Classifier) liegt mit einer Genauigkeit von circa 85.7% zwischen den beiden anderen Modellen. Basierend auf der besten mittleren Kreuzvalidierungs-Genauigkeit, hat der RandomForestClassifier am besten abgeschnitten, gefolgt vom SVC. Die LogisticRegression zeigte die geringste Leistung der drei verglichenen Modelltypen für die zugrundeliegende Aufgabe und den Datensatz.
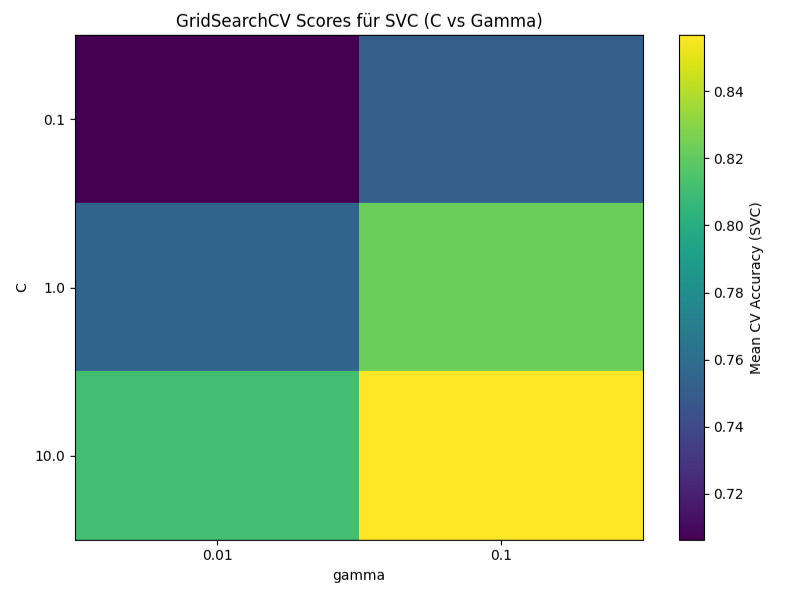
Das Bild visualisiert die Ergebnisse der mittleren Kreuzvalidierungs-Genauigkeit (Mean CV Accuracy) in Abhängigkeit von den Hyperparametern C und Gamma. Jede Zelle in der Heatmap repräsentiert eine Kombination dieser beiden Hyperparameter. Die Farbe der Zelle gibt die erreichte mittlere Kreuzvalidierungs-Genauigkeit für diese Kombination an. Dunkle Farben deuten auf eine niedrigere Genauigkeit hin, während helle Farbe eine höhere Genauigkeit anzeigen.
Aus der Heatmap lässt sich ablesen, dass die höchste Genauigkeit bei der Kombination von C = 10.0 und Gamma = 0.1 erzielt wird. Im Gegensatz dazu wird die niedrigste Genauigkeit bei C = 0.1 und Gamma = 0.01 beobachtet. Generell ist ein Trend erkennbar, dass höhere Werte für C und der höhere Wert für Gamma (0.1) innerhalb der getesteten Bereiche zu besseren Ergebnissen führen.
Das Beispiel demonstriert einen Workflow, der die Hyperparameter-Optimierung und den Modellvergleich mittels Kreuzvalidierung innerhalb einer Pipeline integriert.
Nach den Importen lädt das Skript den „Covertype“-Datensatz. Da dieser Datensatz recht umfangreich ist und die Berechnung mit GridSearchCV zeitaufwendig sein kann, wird für Demonstrationszwecke nur eine Untermenge der ersten 30.000 Datenpunkte verwendet. Diese Untermenge wird dann mittels train_test_split in einen Trainingsdatensatz und einen Testdatensatz aufgeteilt, wobei 30% der Daten für den Testdatensatz reserviert werden und die Aufteilung stratifiziert nach den Klassenlabels erfolgt, um sicherzustellen, dass die Klassenverteilung in beiden Sätzen ähnlich ist.
Anschließend wird eine Basis-Pipeline, base_pipeline, definiert. Diese Pipeline besteht aus zwei Schritten: einem StandardScaler der die Merkmalsdaten transformiert, indem er den Mittelwert entfernt und auf Einheitsvarianz skaliert, und einem Platzhalter-Klassifikator, der initial als SVC-Instanz mit dem Namen ‚classifier‘ definiert ist. Dieser ‚classifier‘-Schritt wird später von GridSearchCV durch die verschiedenen zu testenden Modelle ersetzt.
Der entscheidende Teil für den Vergleich verschiedener Modelle und deren Hyperparameter ist die Definition des param_grid. Jedes Dictionary in dieser Liste repräsentiert einen Satz von Parametern für einen bestimmten Classifier. Das erste Dictionary konfiguriert einen SVC und gibt Suchräume für dessen Hyperparameter C, gamma und kernel an. Das zweite Dictionary richtet einen RandomForestClassifier ein, für den n_estimators , max_depth und min_samples_split optimiert werden sollen. Das dritte Dictionary definiert die Parameter für eine LogisticRegression, wobei C und penalty variiert werden und der ‚liblinear‚-Solver verwendet wird.
Das GridSearchCV-Objekt wird wird mit base_pipeline als zu optimierenden estimator, dem param_grid mit den zu durchsuchenden Parameterräumen und Modelltypen, der Anzahl der Kreuzvalidierungs-Folds (cv=3 für schnellere Ausführung), der Bewertungsmetrik (scoring=’accuracy‘) und die Einstellung refit=True, die sicherstellt, dass das beste gefundene Modell am Ende auf den gesamten Trainingsdaten neu trainiert wird instanziiert.
Der Aufruf von grid_search.fit(X_train, y_train) startet den Optimierungsprozess. GridSearchCV iteriert durch jede Kombination von Modelltyp und Hyperparametern, die im param_grid spezifiziert sind. Für jede dieser Kombinationen wird eine 3-fache Kreuzvalidierung auf den Trainingsdaten durchgeführt. Innerhalb jedes Folds der Kreuzvalidierung wird die gesamte Pipeline auf den Trainingsdaten des Folds angepasst. Der Scaler lernt seine Parameter (Mittelwert, Standardabweichung) nur aus den Trainingsdaten des aktuellen Folds und transformiert diese. Anschließend wird der Klassifikator mit den transformierten Daten trainiert. Die Leistung wird dann auf den Validierungsdaten des Folds bewertet, die ebenfalls mit dem auf den Trainingsdaten des Folds angepassten Scaler transformiert werden.
Die Leistung wird anschließend auf dem separaten Testdatensatz evaluiert. Vorhersagen werden für die Testdaten generiert, und die Genauigkeit (accuracy_score) sowie ein detaillierter classification_report (mit Precision, Recall, F1-Score pro Klasse) werden ausgegeben, um eine Bewertung der Fähigkeit des Modells zu erhalten.
